
WGCNA+Cytoscape:揭示基因网络的深层联系
在生物信息学的研究中,WGCNA(加权基因共表达网络分析)作为一种强大的工具,广泛应用于基因表达数据分析,帮助我们理解复杂的基因调控机制和生物学过程。
01
WGCNA的研究目的
WGCNA的主要目的是通过分析基因的协同表达模式,寻找潜在的基因模块(module),并探索这些模块与研究对象的表型之间的关联关系。此外,WGCNA还可以揭示网络中的核心基因,这些基因可能在特定生物学过程或疾病中发挥关键作用。
02
WGCNA的应用范围
WGCNA适用于复杂的数据模式,尤其推荐在样本数量较大的情况下使用(一般建议至少5组或15个样本)。以下是一些典型的应用场景:
不同器官或组织类型的发育调控
同一组织不同发育阶段的调控
非生物胁迫在不同时间点的应答
病原菌侵染后的时序应答
03
结合Cytoscape可视化分析
通过将WGCNA的分析结果与Cytoscape结合,就可以更直观地展示基因之间的相互关系、模块结构及其与表型的关联。这种可视化方法不仅增强了结果的解释性,还帮助我们发现潜在的关键基因和网络。
下面主要介绍一下使用过程中比较关键的点。
WGCNA主要内容如下:
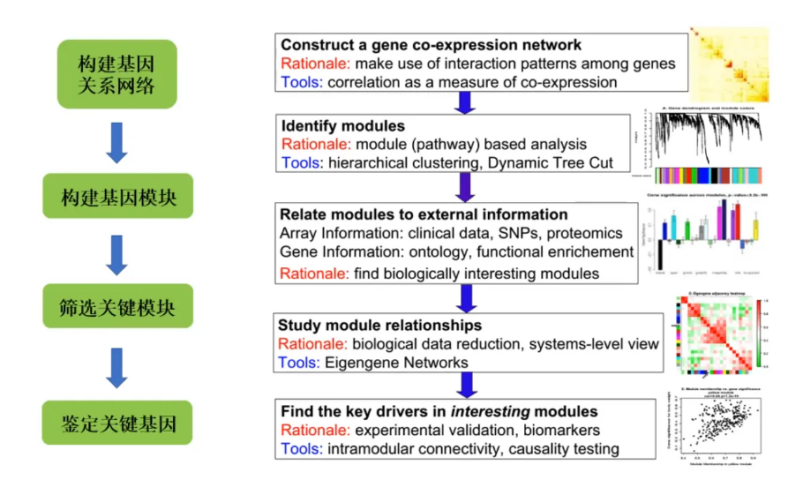
主要影响构建基因关系网络的参数有标红的两个
minModulesize:每个模块最少的基因数量
mergecutHeight:模块融合的阈值,值越大,模块越少
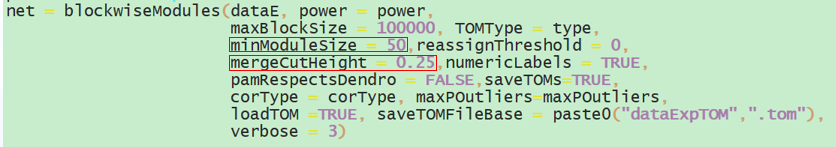
此函数挑选power值,一般选择SFT.R.sq大于0.8的power,下图选择16
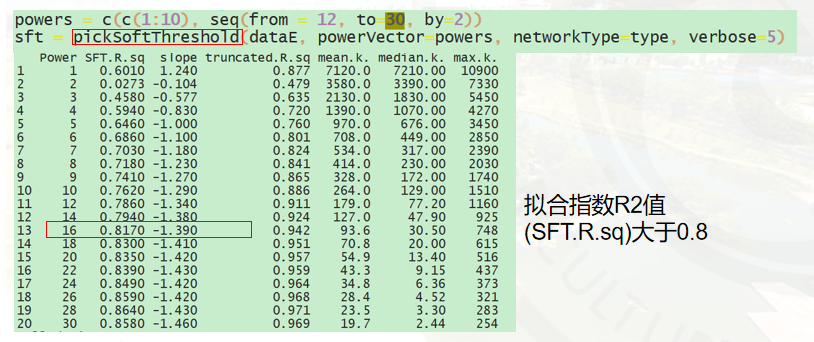
如果没有合适power,可以根据type类型选择。
unsigned:不区分正相关和负相关,官方推荐power=6
signed:区分正负相关,官方推荐power=12
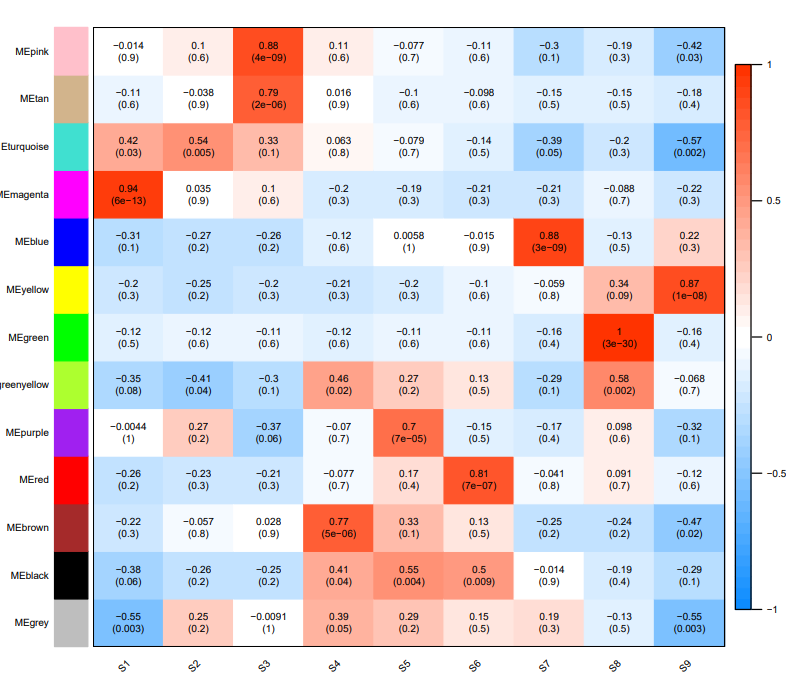
之后将模块与表型或样本关联(以下示例为模块与样本关联)
输入文件参考格式

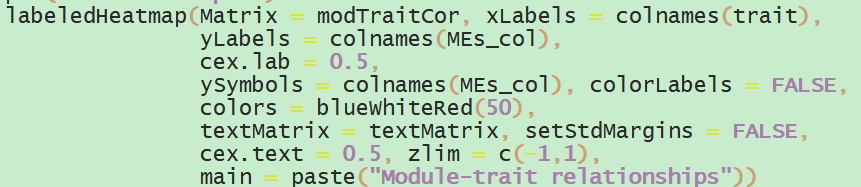
通过绘图得到的网络结果如下
通过模块确定关键基因
dataKME为每个基因与模块的相关性
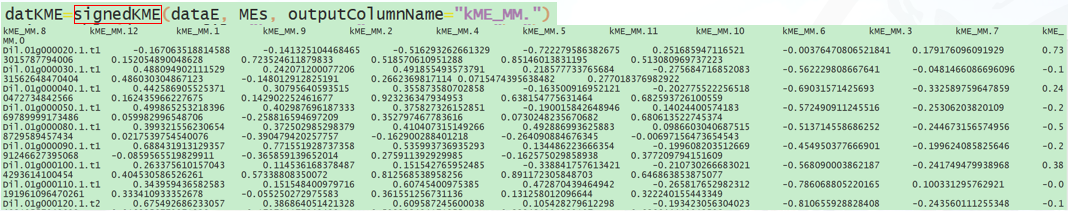
一般筛选与模块相关性大于0.8的基因

也可以提取指定模块大于指定weight的所有基因关系

得到的表就是如下格式,这下可以直接导入到Cytoscape开始绘图啦

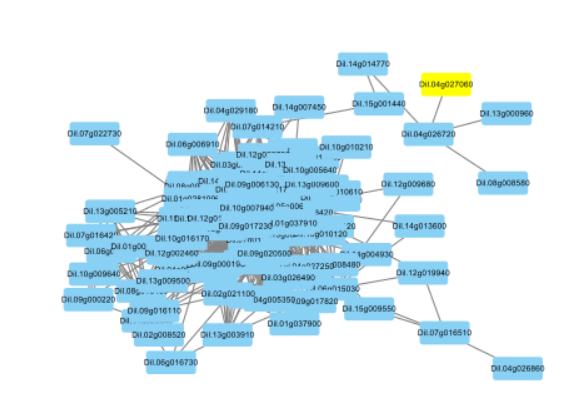
筛选合适数量的关系的导入Cytoscape(筛选weight)(一般不超过2000行,不然太杂乱)
这时的基因名可能还是不利于观察,所以可以导入其他数据库的功能注释信息
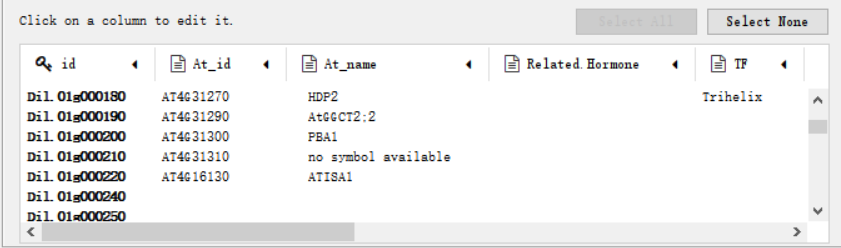
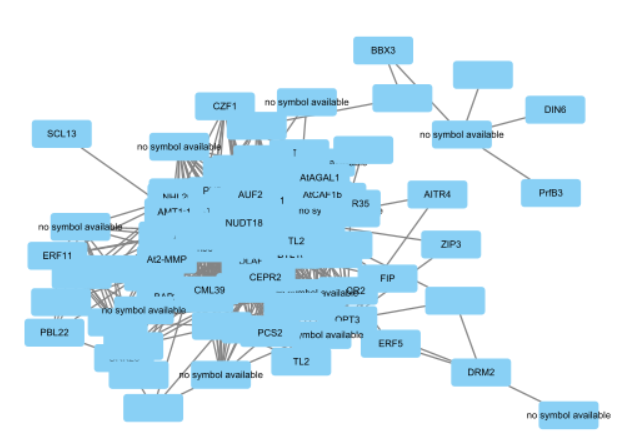
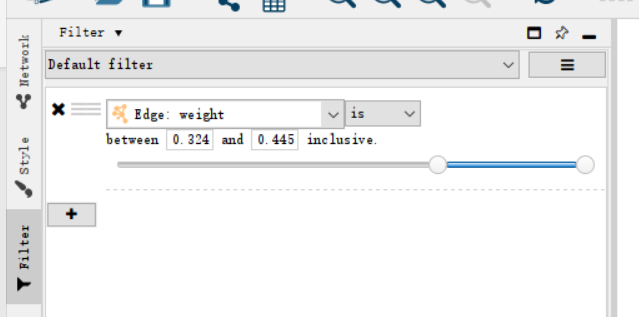
之后再在Style进行一系列的美化,那就可以得到一张较为美丽共表达网络图啦!









