
文章标题:The unique gut microbiome of giant pandas involved in protein metabolism contributes to the host’s dietary adaption to bamboo
发表期刊:Microbiome
影响因子:15.5
组学技术:宏基因组,宏转录组,非靶向代谢组
大熊猫(Ailuropoda melanoleuca)是全球野生动物保护的旗舰物种,为食肉目熊科动物。虽然大熊猫具有典型的食肉性胃肠道特征,但其饮食主要由高纤维的竹子组成,而大熊猫的基因组缺乏消化纤维素和半纤维素所需的酶,研究人员认为肠道微生物在大熊猫健康和营养中可能发挥了重要作用。
本研究通过二代测序(Illumina HiSeq)和三代测序技术(Oxford Nanopore Technologies, ONT),对98只大熊猫的粪便样本进行了深度测序和组装(图1),构建了较为完整的大熊猫肠道微生物基因组(metagenome-assembled genomes, MAGs)。作者利用宏基因组和宏转录组测序技术,将大熊猫肠道微生物与食肉动物、食草动物和杂食动物的肠道微生物进行比较,发现大熊猫肠道微生物的氨基酸代谢显著不同;进一步分析发现非解乳糖链球菌(Streptococcus alactolyticus)可能是大熊猫肠道微生物参与氨基酸代谢的关键微生物,小鼠灌胃实验证明非解乳糖链球菌显著提高小鼠肠道内容物中必需氨基酸的代谢水平。
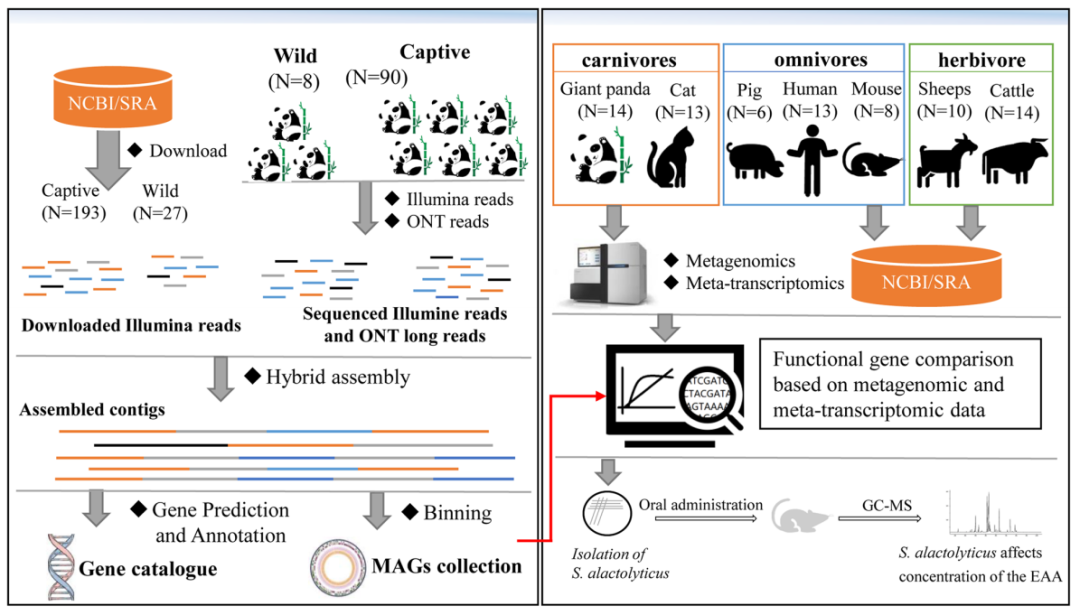
图1 实验设计示意图
1、利用宏基因组组装的大熊猫肠道微生物基因组
为了构建高质量的MAGs,作者首先利用 HiSeq和ONT技术对98只大熊猫的粪便样本进行了深度测序和组装分析(图1)。最终构建出610个中高质量的微生物MAGs,生成了一个由408个MAGs组成的大熊猫非冗余肠道微生物基因组草图(图2 A),其中有148个为高质量MAGs(图2 B)。408个MAGs被分类为细菌界10个门、59个科(图2 C)。其中门水平中厚壁菌门相对丰度最高,属水平中链球菌属相对丰度最高(图3)。在MAGs水平上,Streptococcus alactolyticus MAG086 相对丰度最高(图4)。
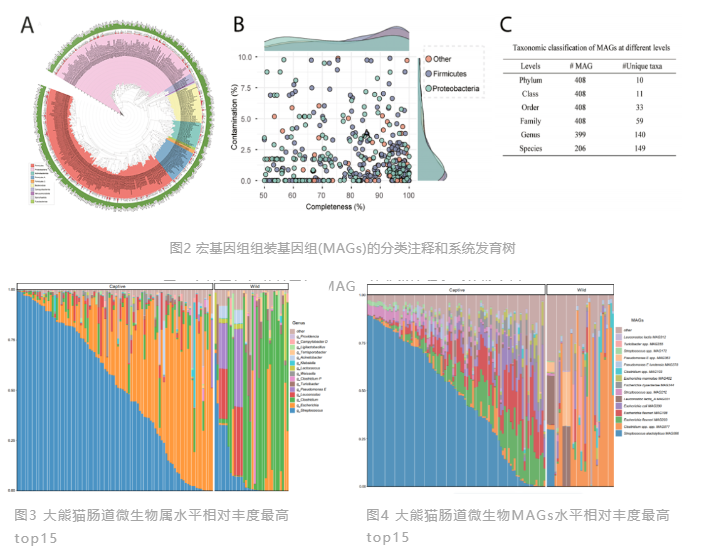
2、大熊猫肠道微生物的宏转录组分析
在全面分析了大熊猫肠道微生物群的MAGs后,作者接下来通过对其中14个圈养大熊猫粪便样本进行宏转录组测序,研究了大熊猫体内活性细菌的基因表达谱。
在种水平上,宏基因组分析显示优势物种为Escherichia flexneri、Streptococcus alactolyticus和Escherichia coli等。相比之下,宏转录分析中优势物种为S. alactolyticus, Escherichia flexneri和Leuconostoc lactis A等(图5)。虽然Escherichia coli在宏基因组中相对丰度排名第三,但是在宏转录组中相对丰度仅排名第六。
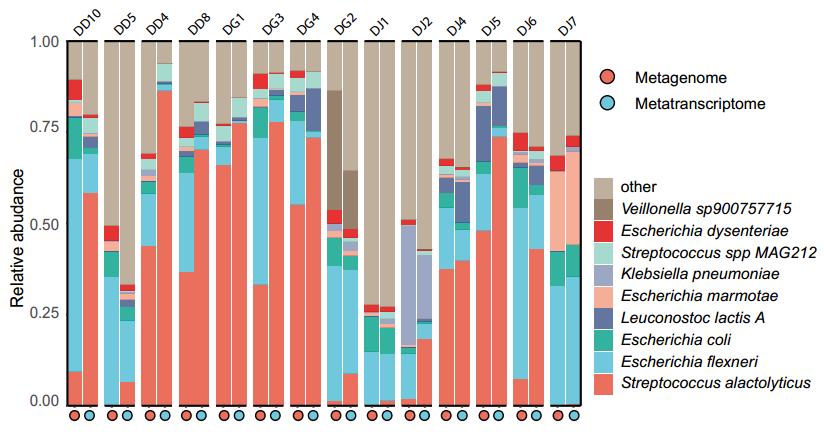
图5 宏基因组和宏转录组中种水平物种相对丰度最高top10
3、大熊猫营养代谢的微生物代谢途径
大熊猫作为食肉动物,却主要以竹子为食。为了探索引起大熊猫这种饮食转变的微生物适应及其功能,作者基于宏基因组和宏转录组研究了主要营养物质(如碳水化合物、氨基酸)的微生物代谢途径,并与三种饮食模式下的哺乳动物,食草动物(牛和羊)、杂食动物(猪、小鼠和人)和食肉动物(猫)进行了比较。
作者首先选择了27个编码纤维素、半纤维素或木质素降解酶的基因来计算不同宿主物种之间的β多样性距离(Bray-Curtis)。宏基因组中,大熊猫中编码植物细胞壁降解代谢途径的微生物基因与猫中更为相似,食肉动物与食草动物、杂食动物在这些基因的组成和丰度上存在显著差异(图6 A)。宏转录组结果也显示为类似的模式(图6 B)。同样,纤维素和半纤维素降解的关键基因,如内切-1,4-β-木聚糖酶、纤维素二糖磷酸化酶和内切纤维素酶的相对丰度和表达量在三种饮食模式下的哺乳动物中也显示出相似的模式,在食肉动物(猫和大熊猫)中基因丰度和表达量均显著低于食草动物(图6 C-E)。这表明大熊猫的肠道微生物不具备帮助宿主利用竹子纤维获得额外能量的潜能。
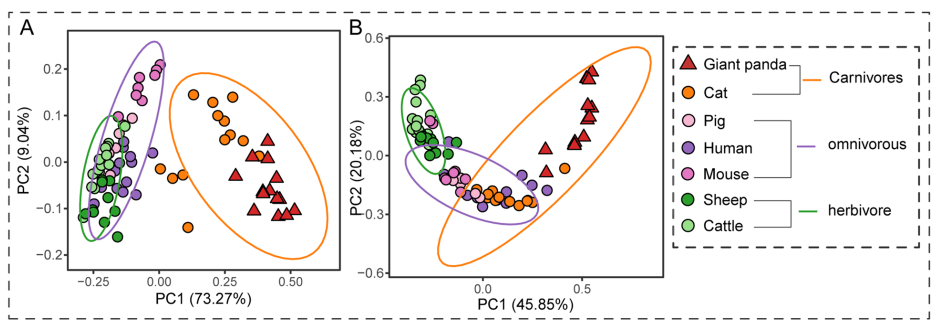
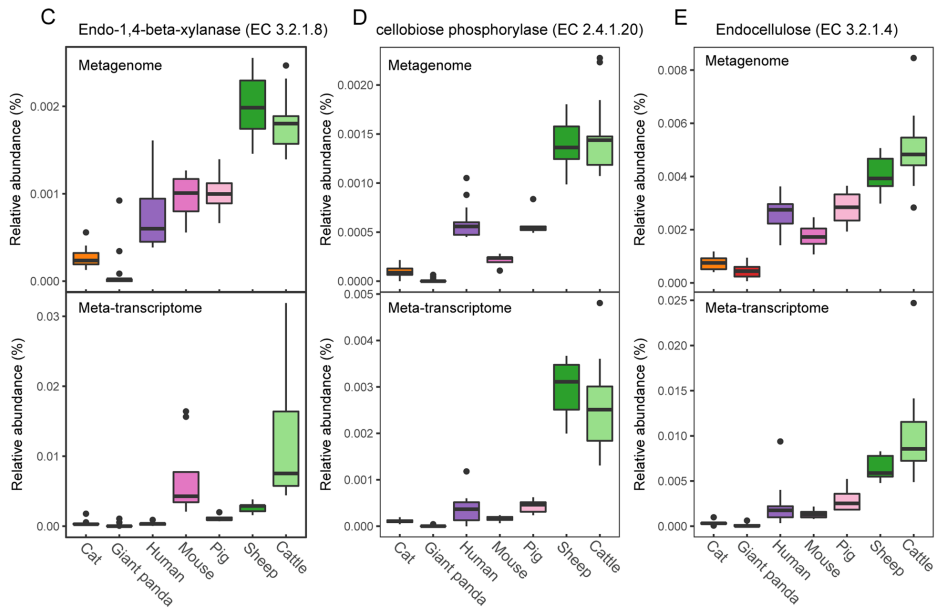
图6 不同宿主肠道微生物中膳食纤维代谢酶相关基因的组成及表达
已有研究表明,竹子中的蛋白质可能是大熊猫的主要能量来源。因此,作者分析了参与氨基酸代谢的基因丰度和表达。宏基因组(图7 A)和宏转录组(图7 B)中大熊猫与其他宿主物种的氨基酸降解功能均存在显著差异,氨基酸生物合成与其他宿主物种间也存在显著差异。
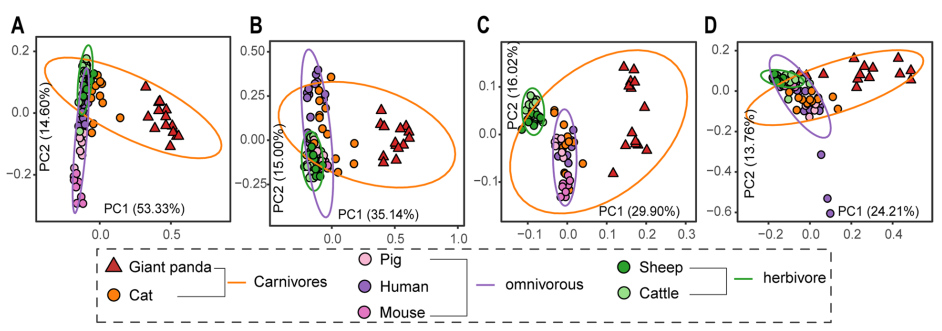
图7 不同宿主肠道微生物氨基酸代谢相关基因的丰度及表达
4、大熊猫肠道微生物群的必需氨基酸生物合成
植物蛋白往往缺少或不足以提供动物所需的某些必需氨基酸。为了探索肠道微生物群是否有助于宿主合成必需氨基酸,作者分析了大熊猫肠道微生物中基因表达水平最高的细菌—非解乳糖链球菌中参与必需氨基酸生物合成的基因丰度。
作者根据非解乳糖链球菌的相对丰度(relative abundance, RA)将圈养大熊猫分为高(RA≥50%, n=28)、中(50%>RA≥25%, n=29)和低(RA<25%, n=33)三组。宏基因组结果中,与必需氨基酸生物合成相关的KEGG通路包括map00290(缬氨酸、亮氨酸和异亮氨酸的生物合成)、map00300(赖氨酸的生物合成)和map00400(苯丙氨酸、酪氨酸和色氨酸的生物合成)。分析发现,高组中map00300 通路(图8 A)和 map00400通路(图8 B)的相对丰度明显高于中组和低组;map00290通路(图8 C)的相对丰度在高组显著高于中组,高组有高于低组的趋势,但无统计学意义。结果表明非解乳糖链球菌可能对大熊猫氨基酸合成有重要的贡献。
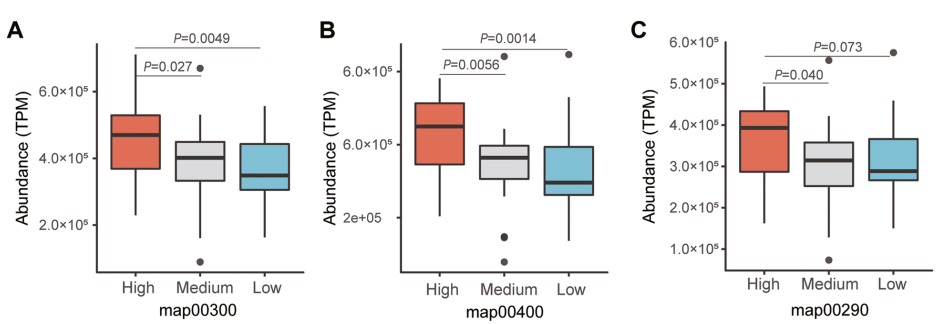
图8 不同非解乳糖链球菌水平下大熊猫必需氨基酸生物合成KEGG通路的相对丰度
为了验证非解乳糖链球菌在肠道必需氨基酸合成中的作用,作者首先从大熊猫粪便中分离出非解乳糖链球菌,然后对低蛋白和正常蛋白饮食的48只BALB/c小鼠菌液分别进行灌胃(低蛋白饮食并灌胃非解乳糖链球菌组LS;低蛋白饮食并灌胃PBS组LC;正常蛋白饮食并灌胃非解乳糖链球菌组NS;正常蛋白饮食灌胃PBS组NC),灌胃结束后三周采集小鼠肠道内容物,通过非靶向代谢组学共检测了9种非必需氨基酸和5种必需氨基酸。对于低蛋白饮食的小鼠,LS组非必需氨基酸和必需氨基酸的总丰度均显著高于LC组,LS组L-缬氨酸和L-酪氨酸的丰度显著高于LC组,而在正常蛋白饮食时,NS组L-谷氨酸的丰度显著高于NC组(图9)。
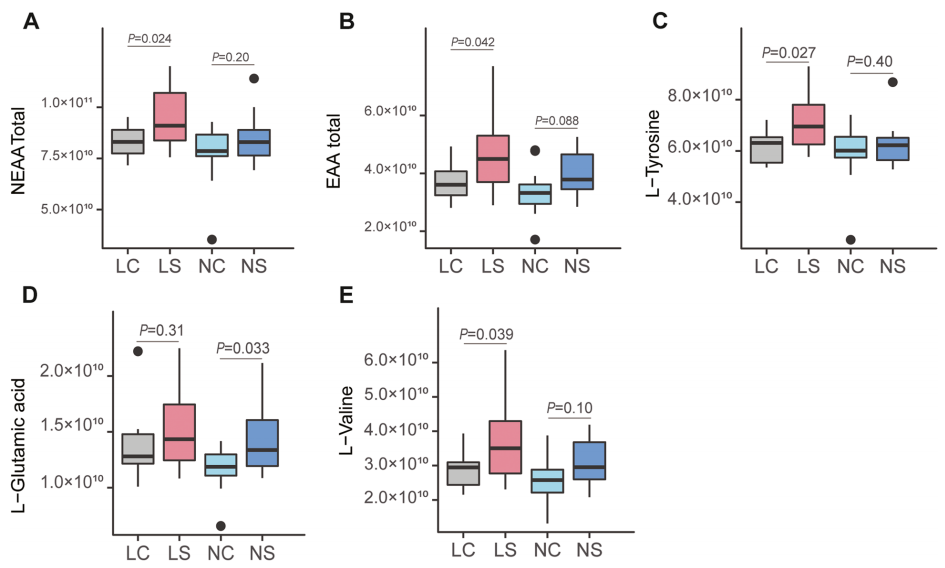
图9 不同处理组小鼠氨基酸丰度的比较
在这项研究中,作者共构建了408个MAGs,这些数据不仅大大改善了我们对熊猫肠道微生物多样性和代谢的看法,同时也为未来的宏基因组学研究提供了数据基础。此外,作者还对一部分样本中的宏基因组和宏转录组进行了配对研究,以区分丰富和活跃的细菌,并确定了参与营养代谢的基因表达谱。
本研究结果表明,大熊猫肠道微生物群在蛋白质代谢过程中发挥着重要作用,其中S. alactolyticus是大熊猫肠道中数量最多的细菌物种,参与了大熊猫肠道的蛋白质代谢。此外,本研究还发现了熊猫肠道微生物组中独特的蛋白质代谢谱,以及他们类似食肉动物短且直的肠道,它们的牙齿、头骨和“伪拇指”对处理竹子所需的进化适应,这也部分解释了大熊猫从肉食转变为食用竹子的饮食结构改变的潜在原因。









